Finding restriction enzyme map |
|
| JEMBOSS 的 Remap 程式可用來尋找核酸序列上的限制酵素 (restriction enzyme) 圖譜。 |
 |
進入Remap程式 |
|
 |
輸入序列 |
|
| |
 |
From database:在 sequence filename 的欄位中輸入資料庫及序列名稱
(或序列的 accession number),可直接由資料庫中叫出序列 (如 embl:eclac)。 |
|
 |
Copy/paste:將已有序列 copy/paste 輸入。 |
|
 |
From local file:將已儲存於個人電腦中的序列檔案,利用拖曳的方式,
由右方的 file manager 中拉到 sequence filename 的欄位內。 |
 |
選擇 restriction enzyme |
|
| |
在 Comma separated enzyme list 的欄位裡輸入 restriction enzyme 的名稱,請使用小寫字母,且須將羅馬數字 I, II, III 以小寫 i, ii, iii 表示,兩個 restriction enzyme 名稱之間以逗號做區分,不需加入空白鍵。 |
| |
例:尋找 ECLAC (E.coli lactose operon with lacI, lacZ, lacY and lacA genes) 所有切位,則在 Comma separated enzyme list 的欄位輸入 all;如在Comma separated enzyme list 的欄位輸入 taqi, acii, fati, bse1i ,則可只尋找這四個限制酵素的切位。 |
| |
|
 |
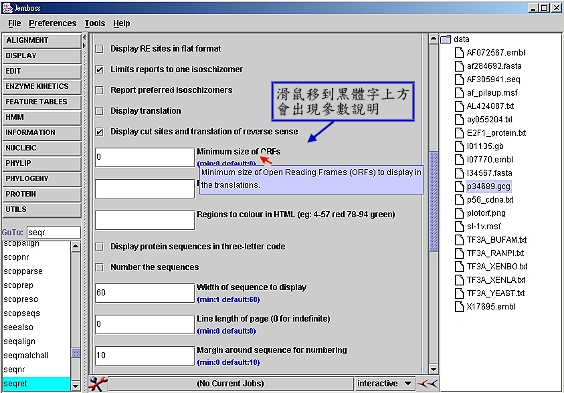
設定參數 |
|
| |
以滑鼠指標移到參數標題的黑體字上,可檢視參數的說明,然後依需要設定。 |
|
 |
 |
選擇Display方式 |
|
| |
Remap預設的 display方式是在序列切點上方標示 restriction enzyme,並在 DNA 序列下方列出對應的蛋白質序列。可依需要勾選或刪除 display的選項。 |
 |
設定advanced options |
|
| |
點選 Advanced Options,頁面往下拉,出現"force single site only cuts", "allow blunt end cutters"等多種選項,可依實驗所需勾選合適的項目。其中也包括 codon table 選項,如果選擇 display translation,此時可依序列來源不同選擇適當的 amino acid 編碼方式,以轉譯出正確的蛋白質序列。 |
 |
按 GO 執行 。 |
|
| |
例:ECLAC所有酵素 (all) 與 TaqI,AciI,FatI,Bse1I四種酵素切點結果: |
| |
|
| |
|
|
|

